MAMUTCELL
Mathématiques multi-échelles pour la dynamique de cellules
Suivi de la propagation d’une colonie
Axe Vivant
Mathématiques
∩ Biologie
Aperçu
Comprendre comment des systèmes vivants s’organisent à grande échelle. Modéliser des systèmes vivants de très grande taille (colonies de micro-organismes, écosystèmes bactériens du sol, réseaux de régulation de gènes, tissus en développement), afin d’appréhender comment l’auto-organisation à l’échelle macroscopique peut émerger d’interactions au niveau microscopique.
Mots clefs
Mathématiques appliquées à la biologie, équations aux dérivées partielles, transport optimal de mesures, processus stochastiques, microbiologie, mouvement collectif, écosystème, expression de gènes, biologie du développement
Coordination
- Vincent Calvez (Mathématiques, Laboratoire de Mathématiques de Bretagne Atlantique (LMBA), UMR 6205, CNRS, Université de Bretagne Occidentale, Université Bretagne Sud)
- Tâm Mignot (Biologie, Laboratoire de Chimie Bactérienne (LCB), UMR 7283, CNRS, Aix-Marseille Université)
Financement
- ≈ 1 million d’euros sur 5 ans
Résumé
Le projet ciblé MAMUTCELL réunit des mathématiciennes et des mathématiciens ainsi que des biologistes autour de questions fondamentales de dynamique cellulaire à des échelles multiples. Les avancées technologiques récentes ont permis de poser un regard nouveau sur les comportements individuels de cellules au sein d’un vaste collectif.
D’une part, il est désormais possible de scruter des trajectoires individuelles de bactéries au sein d’une foule dense, et par là-même d’interroger les règles de décision individuelles qui conduisent à l’émergence de mouvements collectifs singuliers (courants cellulaires, vagues synchronisées).
D’autre part, il est possible de mesurer la diversité dans l’expression des gènes au cours des différentes étapes de différentiation d’une population de cellules, et par suite de mesurer la variabilité des processus intracellulaires qui opèrent cette différentiation.
Ces progrès spectaculaires appellent des développements mathématiques dans la modélisation et l’analyse des populations de cellules et de leur hétérogénéité, qu’elle soit spatiale ou génétique. Des cadres formels ont émergés naturellement, que ce soit les modèles cinétiques à l’échelle mesoscopique, ou des modèles individu-centrés à l’échelle microscopique pour les dynamiques spatiales, ou encore les flots de mesures pour les dynamiques génétiques. Ce projet a pour but de faire progresser la modélisation et l’analyse au sein de ces cadres formels, tout en gardant un ancrage fort dans la biologie, afin d’améliorer la compréhension de la complexité du vivant au travers des échelles, de l’individu au collectif.
Ce projet propose également d’ouvrir des pistes de recherche ambitieuses à l’interface mathématiques-biologie dans des environnements complexes, que ce soit l’étude de bactéries prédatrices au sein d’un écosystème, ou bien le suivi de la différentiation de cellules au sein d’un tissu en développement
- Obtenir des conclusions biologiques robustes à partir de modèles et d’analyses mathématiques rationnelles
- Donner du sens à des données massives issues de microscopie ou de séquençage ARN
- Analyse multi-échelle, dérivation de modèles macroscopiques bien adaptés, processus de Markov déterministe par morceaux, transport optimal non équilibré
- Données de microscopie à haute résolution spatiale, données métagénomiques, expression de gènes en cellule unique
- Développer de meilleurs outils mathématiques pour comprendre la complexité et les dynamiques du vivant
- Développer des méthodes bien calibrées pour étudier des mouvements de foule (ici, à l’échelle des micro-organismes)
- Participer à une synergie toujours plus forte entre disciplines
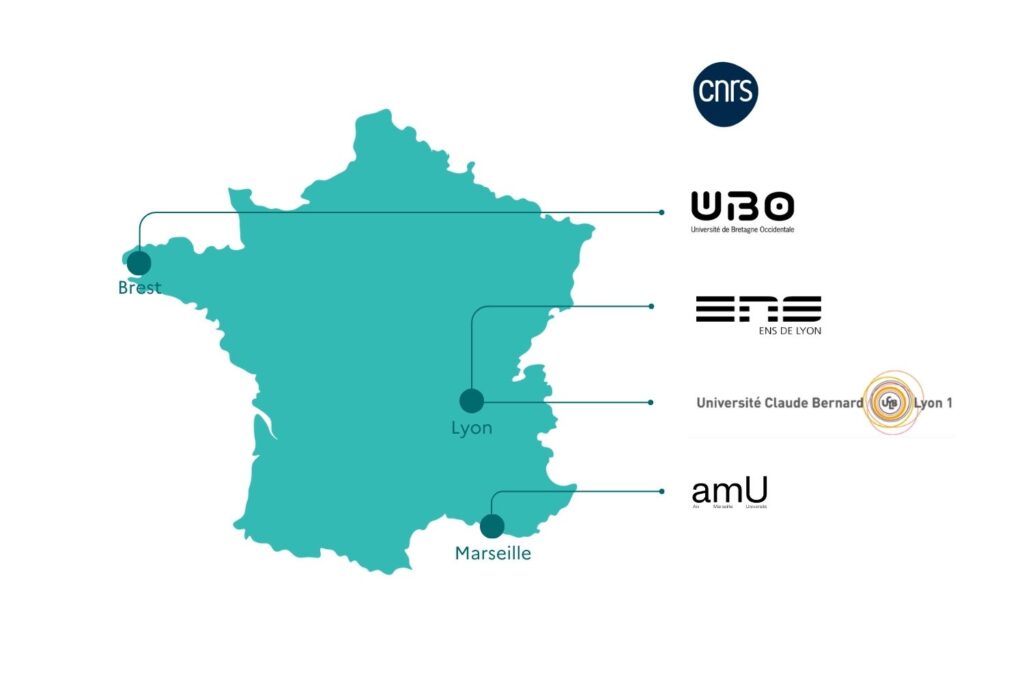
Consortium
- Laboratoire de Mathématiques de Bretagne Atlantique (LMBA), UMR 6205, CNRS, Université de Bretagne Occidentale, Université Bretagne Sud (Brest)
- Laboratoire de Chimie Bactérienne (LCB), UMR 7283, CNRS, Aix-Marseille Université (Marseille)
- Institut Camille Jordan (ICJ), UMR 5208, CNRS, École Centrale de Lyon, INSA Lyon, Université Claude Bernard Lyon 1, Université Jean Monnet (Lyon)
- Laboratoire de Biologie et Modélisation de la Cellule (LBMC), UMR 5239, CNRS, ENS de Lyon (Lyon)
Recrutements envisagés
- 5 contrats de thèse (contrat de 3 ans) ou de post-doctorat (contrat de 2 ans)
Plus de projets